
Mikroskopijny najeźdźca we krwi
 Słowa „sepsa" lub „posocznica" co rusz pojawiają się w nagłówkach gazet lub padają z ust dziennikarzy, wprawiając w drżenie rzesze czytelników, słuchaczy i telewidzów. Oba terminy oznaczają to samo, czyli zakażenie obejmujące cały organizm i rozprzestrzeniające się we krwi.
Słowa „sepsa" lub „posocznica" co rusz pojawiają się w nagłówkach gazet lub padają z ust dziennikarzy, wprawiając w drżenie rzesze czytelników, słuchaczy i telewidzów. Oba terminy oznaczają to samo, czyli zakażenie obejmujące cały organizm i rozprzestrzeniające się we krwi.Nie oznacza to jednak, że sepsa grozi każdemu z nas. Codziennie organizm człowieka jest wystawiony na działanie milionów drobnoustrojów, ale nie prowadzi to do ogólnoustrojowej infekcji, ponieważ wciąż działa nasz system immunologiczny (odpornościowy). Sepsa jest wywoływana przez bakterie lub (rzadziej) grzyby chorobotwórcze. Dochodzi do niej w przypadku, gdy organizm człowieka jest osłabiony (np. wyniszczenie chorobą nowotworową, zakażenie wirusem HIV, przewlekłe choroby, jak np. nieleczona cukrzyca itp.) i nie jest w stanie skutecznie obronić się przed drobnoustrojami, które przedostają się do krwi, na przykład podczas zabiegu operacyjnego lub po przebytej infekcji. Niestety, sporo pacjentów umiera z powodu sepsy, mimo leczenia na oddziałach intensywnej terapii i zastosowania arsenału antybiotyków. W USA co roku na sepsę zapada ok. 750 tysięcy osób i jest ona przyczyną ponad 215 tysięcy zgonów. W Unii Europejskiej z powodu sepsy umiera rocznie 146 tysięcy pacjentów.
Niepewne testy
Aby dać pacjentowi szansę na pokonanie zakażenia, konieczne jest szybkie i skuteczne stwierdzenie, czy i jaki drobnoustrój jest przyczyną sepsy. Taka informacja pozwala lekarzowi na wdrożenie prawidłowej terapii lekami przeciwdrobnoustrojowymi i zwiększa szansę na przeżycie. Od wielu lat jedyną dostępną metodą diagnostyczną są posiewy próbek krwi celem wyhodowania z nich drobnoustrojów (bakterii lub grzybów) będących przyczyną zakażenia. Niestety, ten sposób postępowania – choć rozpowszechniony na całym świecie – nie pozwala na wykrycie mikrobiologicznego drobnoustroju patogennego w nawet 60% przypadków, mimo że mamy do czynienia z sepsą. Ograniczeniem tego rodzaju analizy jest czułość metody hodowlanej. Kolejny problem stanowi czas potrzebny na uzyskanie wyniku z laboratorium – w skrajnym wypadku trzeba czekać na rezultat nawet do tygodnia, licząc od momentu pobrania próbek, a to zdecydowanie zbyt długo dla umierającego.
Szansą na wyeliminowanie opisanych trudności jest zastosowanie w diagnostyce metod opartych na wykrywaniu DNA lub RNA drobnoustrojów bezpośrednio we krwi u pacjentów z objawami sepsy. Na rynku dostępne są obecnie zaledwie trzy komercyjne testy wykorzystujące metody genetyczne, które pozwalają na potwierdzenie obecności we krwi kilku lub kilkunastu najbardziej prawdopodobnych gatunków bakterii czy grzybów. Nie wyczerpuje to potrzeb związanych z diagnostyką zakażenia dlatego, że te testy są zaprojektowane w ten sposób, by wykrywać panel najczęściej występujących drobnoustrojów we krwi. Wynik negatywny nie jest więc gwarantem, że niczego tam rzeczywiście nie ma. Wciąż istnieje zatem potrzeba prowadzenia prac badawczych nad tym problemem.
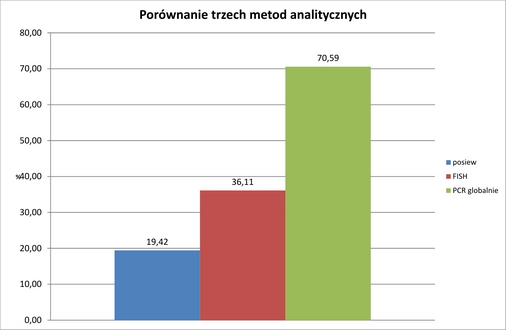
Porównanie skuteczności wykrywania drobnoustrojów.
Badania przeprowadzono na 102 próbkach krwi pobranych
od pacjentów z objawami sepsy
Tylko sześć godzin
W Katedrze Mikrobiologii Collegium Medicum Uniwersytetu Jagiellońskiego od 2010 roku prowadzone są badania zmierzające do opracowania nowatorskich metod diagnostycznych stosowanych w zakażeniach drobnoustrojami. Projekt ten finansowany jest z grantu Narodowego Centrum Nauki (NCN). „W chwili obecnej już wiemy, jak wykrywać bakterie oraz grzyby bezpośrednio w próbkach krwi pacjentów z objawami sepsy i to bez konieczności prowadzenia czasochłonnej hodowli. Opracowana metoda pozwala wykryć zakażenie we krwi w czasie do sześciu godzin od momentu pobrania próbki krwi. Dysponujemy dwiema odmianami metod: jedna pozwala na zobrazowanie komórek drobnoustrojów w próbce krwi przy wykorzystaniu mikroskopu fluorescencyjnego – FISH (ang. Fluorescent In Situ Hybridization), a druga, tzw. PCR (ang. Polymerase Chain Reaction), bazuje na amplifikacji (namnażaniu) markerowych fragmentów DNA drobnoustrojów w materiale genetycznym wyizolowanym z próbki krwi pacjenta" – informuje dr Tomasz Gosiewski, koordynujący badania nad szybkimi sposobami diagnostyki sepsy.
Czułość metody FISH pozwala potwierdzić obecność komórek mikroorganizmów we krwi, jeśli jest ich przynajmniej 1000 w 1 ml krwi, natomiast PCR pozwala na detekcję na poziomie 100 komórek/ml. Koszt przeprowadzenia badania z wykorzystaniem metody FISH jest ok. trzykrotnie niższy niż w przypadku metody PCR. Wynika to ze specyfiki analizy niewymagającej używania enzymów do namnażania DNA drobnoustrojów w probówce, co jest konieczne do potwierdzenia ich obecności. Obydwa sposoby umożliwiają różnicowanie wykrywanych drobnoustrojów na bakterie Gram ujemne, Gram dodatnie, grzyby drożdżowe i grzyby pleśniowe, a więc na grupy obejmujące wszystkie patogenne gatunki bakterii oraz grzybów. Informacja na ten temat daje lekarzowi możliwość zastosowania bardziej precyzyjnego leczenia za pomocą antybiotyków.
Opracowane metody zostały opisane w specjalistycznych anglojęzycznych czasopismach (np. w „Current Microbiology" 2014) oraz zaprezentowane na krajowych i międzynarodowych konferencjach naukowych (np. „23rd European Congress of Clinical Microbiology and Infectious Diseases", Germany, Berlin 2013).
Analiza PCR została ponadto zgłoszona do ochrony patentowej w Polsce i za granicą. Możliwe jest dalsze rozwijanie metod, aby umożliwić typowanie konkretnych gatunków bakterii i grzybów oraz wykrywać oporność na antybiotyki, co pozwoli już na starcie eliminować nieskuteczne leczenie.
Projektor Jagielloński 2, "Sepsa, czyli mikroskopijny najeźdźca we krwi", www.projektor.uj.edu.pl
Tagi: krew, sepsa, posocznica
wstecz Podziel się ze znajomymi



Najdokładniejsze systemy satelitarnego transferu czasu
Nie zawsze zegar atomowy działa lepiej niż kwarcowy.

Ponad połowa chorych z SARS-CoV2 cierpi na długi covid
Przez długi czas może mieć takie objawy jak zmęczenie.



Uniwersytet Warszawski będzie kształcić kadry dla energetyki jądrowej
Przekazał Wydział Fizyki UW.










Recenzje