- Biochemia
- Biofizyka
- Biologia
- Biologia molekularna
- Biotechnologia
- Chemia
- Chemia analityczna
- Chemia nieorganiczna
- Chemia fizyczna
- Chemia organiczna
- Diagnostyka medyczna
- Ekologia
- Farmakologia
- Fizyka
- Inżynieria środowiskowa
- Medycyna
- Mikrobiologia
- Technologia chemiczna
- Zarządzanie projektami
- Badania kliniczne i przedkliniczne
Aptamery nukleinowe jako wszechstronne detektory biologii molekularnej
Aptamery nukleinowe są jednoniciowymi oligonukleotydami kwasu rybonukleinowego (RNA) lub deoksyrybonukleinowego (DNA), które wykazują wysokie powinowactwo i specyficzność wiązania do ściśle określonych biomolekuł lub cząsteczek nieorganicznych. Nazwa aptamer wywodzi się od łacińskiego słowa „aptus”, które oznacza „przyczepiony, dopasowany” i dobrze odzwierciedla charakter tych związków. Aptamer pełni rolę liganda, wiążącego się bardzo silnie, świadczy o tym nano- lub pikomolowa stała dysocjacji (Kd) tak powstałych kompleksów aptamer-cząsteczka docelowa, będąca miarą stabilności kompleksu. Od 2002 roku wzrasta światowe zainteresowanie ich wytwarzaniem oraz praktycznymi zastosowaniami zarówno w badaniach naukowych, jak i w medycynie. Aptamery są obiecującym narzędziem do zwalczania niektórych chorób wirusowych, nowotworowych, autoimmunizacyjnych oraz chorób układu krążenia.
Wprowadzenie
Monomerem kwasu nukleinowego jest nukleotyd, który składa się z nukleozydu czyli cząsteczki pentozy - cukru (dla RNA rybozy, dla DNA deoksyrybozy), do której przyłączona jest wiązaniem N-glikozydowym przy pierwszym atomie węgla - zasada azotowa (purynowa lub pirymidynowa) oraz z reszty fosforanowej, która w nukleotydzie łączy trzeci oraz piąty atom węgla dwóch sąsiednich pentoz polimeru. Zatem między nukleotydami występuje wiązania fosfodiestrowe. Zasadami są adenina, guanina, cytozyna oraz uracyl (w RNA) lub tymina (w DNA).
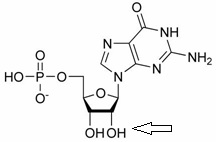
Aptamery RNA i DNA wykazują nieco odmienne właściwości. Kwasy rybonukleinowe tworzą najczęściej bardziej złożone struktury niż DNA, na co pozwala im obecność grup 2`- OH w pierścieniu rybozy biorących udział w tworzeniu wiązań wodorowych w obrębie aptameru lub między aptamerem a cząsteczką docelową (Rys. 1).
Macugen
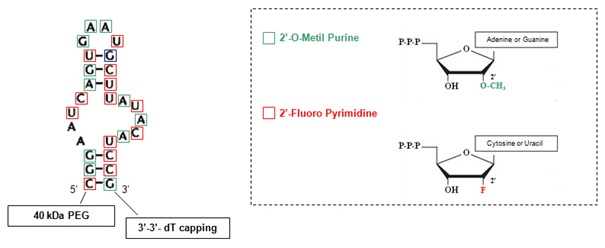
Pierwszy lek na bazie aptameru zatwierdzony przez FDA (ang. US Food and Drug Administration), który ukazał się w ostatnich latach, służy w leczeniu zwyrodnienia plamki żółtej (AMD). Nosi on nazwę Macugen®, a oferowany jest przez OSI Pharmaceuticals. Aptamer wiąże i antagonizuje działanie białka VEGF-165, izoformy VEGF zaangażowanej w patologicznej neowaskularyzacji oka. W celu poprawy farmakodynamicznych i farmakokinetycznych właściwości tego 28-merowego aptameru zmodyfikowano go poprzez wprowadzenie na węglu 2' pirymidyn atomów fluoru (-F) oraz grup metoksylowych (-OMe) na węglu 2' w purynach (Rys. 2). Zmodyfikowany aptamer związano z cząsteczkami PEG (glikol polietylenowy), co zwiększyło czas przebywania wewnątrz ciała szklistego.
Mechanizm rozpoznawania cząsteczki docelowej
Bardzo silne oddziaływanie z rozpoznawaną molekułą, porównywalne z oddziaływaniem przeciwciała monoklonalnego z antygenem, jest wynikiem niezwykłego dopasowania struktury przestrzennej aptameru do budowy swego „celu”.
Jednoniciowe kwasy nukleinowe mogą fałdować się, tworząc obok wielu fragmentów helikalnych złożone struktury drugo- i trzeciorzędowe (Rys. 3). Strukturę aptamerów stabilizują różne wiązania chemiczne: wiązania wodorowe, oddziaływania elektrostatyczne oraz oddziaływania van der Waalsa (dipol-dipol). Powstałe motywy strukturalne umożliwiają trwałe „wpasowanie się” liganda aptamerowego do danej biocząsteczki lub odwrotnie - jeśli oligonukleotyd jest większy od swego celu. W trakcie rozpoznania celu molekularnego biorą udział zarówno elementy aptameru, jak też i badanej molekuły. Co ciekawe, fragment wiążący zwykle nie jest dłuższy od piętnastu nukleotydów.
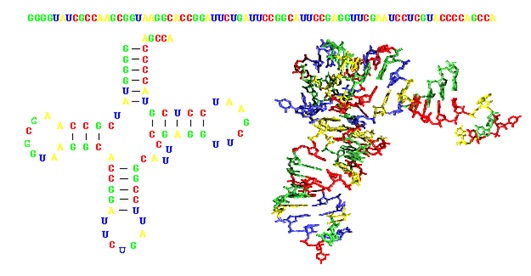
W zależności od rodzaju cząsteczki docelowej rozpoznanie molekularne może następować pomiędzy różnymi motywami struktur drugo- i trzeciorzędowych np. związki drobnocząsteczkowe takie jak aminokwasy lub ATP (adenozynotrójfosforan) dołączają się do aptameru wykorzystując do interakcji następujące rodzaje motywów: pseudowęzły (ang. pseudoknots), wybrzuszenia dwustronne (ang. internal loops), wybrzuszenia jednostronne (ang. bulges). W przypadku dużych cząsteczek, to aptamery inkorporują w strukturę docelowej molekuły wykorzystując różnego rodzaju oddziaływania chemiczne: oddziaływania niekowalencyjne, wiązania wodorowe, siły Van der Waalsa, oddziaływania elektrostatyczne oraz oddziaływania hydrofobowe.
Rodzaje cząsteczek rozpoznawanych przez aptamery
Aptamery nukleinowe rozpoznają związki wielkocząsteczkowe, takie jak enzymy, białka regulatorowe, czynniki wzrostu, przeciwciała mono- i poliklonalne, lektyny oraz niskocząsteczkowe witaminy, antybiotyki, barwniki organiczne, aminokwasy, peptydy, nukleotydy a nawet niektóre jony metali. W warunkach naturalnych wiele spośród tych molekularnych „celów” w ogóle nie oddziałuje z kwasami nukleinowymi.
Właściwości niemodyfikowanych aptamerów
Niemodyfikowane aptamery są szybko usuwane z krwi. Przy czym ich okres półtrwania zawiera się w przedziale od kilku minut do godziny, głównie ze względu na degradację w wyniku działania enzymów nukleolitycznych oraz oczyszczanie przez nerki. Ten szybki klirens może być korzystny w takich zastosowaniach jak w diagnostycznym obrazowaniu in vivo.
Wytwarzanie aptamerów nukleinowych - proces SELEX
Najpowszechniej stosowaną metodą otrzymywania aptamerów RNA i DNA o pożądanych właściwościach fizycznych i chemicznych jest ich selekcja w warunkach in vitro. Proces wytwarzania aptamerów nukleinowych i izolowania tych z nich, które specyficznie wiążą się do określonej molekuły przedstawiono już 1990 roku. Metodę nazwano w skrócie SELEX (ang. Systematic Evolution of Ligands by Exponential Enrichment). Polega ona na zidentyfikowaniu i wybiórczej amplifikacji oligonukleotydów o oczekiwanej charakterystyce spośród licznej puli przypadkowych sekwencji, w sposób analogiczny do naturalnych mechanizmów ewolucyjnych.
W metodzie SELEX można wyróżnić trzy główne etapy, a mianowicie utworzenie kombinatorycznej biblioteki RNA lub DNA, selekcję oligonukleotydów oraz powielenie wyselekcjonowanych aptamerów metodami biologii molekularnej (Rys. 4).
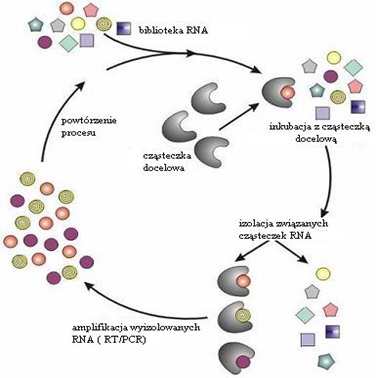
Pierwszym etapem procesu jest utworzenie kombinatorycznej biblioteki DNA lub RNA na drodze syntezy chemicznej - bibliotekę tworzy 1014-1015 makrocząsteczek o określonej długości i różnych „kształtach”. Każdy z oligomerów w takiej bibliotece posiada fragment środkowy o zupełnie przypadkowej sekwencji nukleotydowej i jest oflankowany od końca 5' i 3' sekwencjami stałymi.
W trakcie syntezy regionu zmiennego stężenia monomerów A, T ( lub U), G i C w mieszaninie reakcyjnej są równomolowe, aby prawdopodobieństwo wystąpienia każdej kombinacji nukleotydów było jednakowe. W przypadku n-nukleotydowego fragmentu daje to 4n różnych kombinacji. Tym samym otrzymuje się pulę oligonukleotydów reprezentujących teoretycznie każdą możliwą sekwencję nukleotydów.
Kolejnym etapem procesu jest selekcja oligonukleotydów specyficznie wiążących daną cząsteczkę. Pulę oligonukleotydów DNA lub RNA poddaje się inkubacji z cząsteczką docelową w określonych warunkach pH, temperatury itp. Cząsteczka docelowa jest zwykle unieruchomiona na błonie nitrocelulozowej. Te z sekwencji, które wiążą się specyficznie do molekuły nie ulegają usunięciu w wyniku następującego płukania i są powielane metodą PCR (lub PCR sprzężonego z odwrotną transkrypcją w przypadku biblioteki RNA). Proces selekcji powtarza się stopniowo zaostrzając warunki reakcji. W czasie każdej z rund i płukań wzrasta udział oligonukleotydów rozpoznających specyficznie cząsteczkę docelową.
Ostatnim etapem procesu jest powielenie wyselekcjonowanych aptamerów. Uzyskane w wyniku kilku lub kilkunastu cykli selekcji oligonukleotydy testuje się pod względem siły wiązania (wyznacza się stałą dysocjacji (Kd) powstałego kompleksu) oraz powinowactwa, a następnie te najlepsze wprowadza do plazmidów i klonuje w komórkach bakteryjnych, po czym sekwencjonuje. Po poznaniu niepowtarzalnej sekwencji nukleotydowej wytwarza się wiele kopii danego aptameru drogą syntezy chemicznej.
Modyfikacje aptamerów
W celu optymalnego wykorzystania aptamerów często modyfikuje się wyselekcjonowane oligonukleotydy, aby zwiększyć ich stabilność, powinowactwo do cząsteczki docelowej lub też umożliwić ich detekcję dostępnymi metodami analitycznymi.
Dimeryzacja wyselekcjonowanych aptamerów pozwala na wielokrotny wzrost powinowactwa liganda do badanej molekuły oraz wzrost trwałości utworzonego kompleksu. Oligonukleotydy po dimeryzacji mają większą liczbę miejsc wiążących, dlatego możliwe jest ich oddziaływanie z więcej niż jedną cząsteczką docelową.
Alkilacja kwasów nukleinowych prowadzi do otrzymania aptamerów zawierających 2`-O-metylopirymidynę. Takie zmodyfikowane aptamery są stabilniejsze i bardziej odporne na ataki enzymów egzonukleolitycznych.
Podstawienie grupy 2`-OH rybozy grupą aminową lub atomami fluoru tysiąckrotne zwiększa odporność na degradację (ciecie) w surowicy krwi przez obecne w niej enzymy.
Wytwarzanie tioaptamerów poprzez modyfikację reszt fosforanowych polegaj na zastąpieniu jednego lub więcej atomów tlenu atomami siarki. Tak zmodyfikowane oligonukleotydy są bardziej odporne na trawienie przez 3`-egzonukleazę.
LNA (ang. Locked Nucleic Acids) polega na wprowadzeniu mostków metylenowych łączących atomy 2'-O i 4'-O w pierścieniu rybozy. Zabieg ten chroni przed degradacją nukleolityczną.
Konstrukcja koliście zamkniętych aptamerów tzw. aptamerów polegająca na ligacji wolnych końców. Kaptamery są znacznie stabilniejsze, a jednocześnie modyfikacja ta nie wpływa na ich zdolności rozpoznawania cząsteczek docelowych.
Tworzenie aptamerów L-DNA i L-RNA (aptamery lustrzane). Wykorzystuje się stereoselektywność enzymów komórkowych wykazujących aktywność tylko w stosunku do enancjomerów występujących w przyrodzie np. rybonukleazy mogą rozpoznać i hydrolizować tylko oligorybonukleotydy oparte na szeregu D-rybozy natomiast nie degradują enancjomerów RNA zawierających L-rybozę.
Znakowanie cząsteczek aptamerów przy pomocy izotopów 3H lub 32P pozwala na monitorowanie kolejnych rund selekcji oraz wykorzystywane jest w diagnostyce obrazowej.
Koniugacja z barwnikami fluorescencyjnymi. W tym celu stosuje się tzw. cząsteczki pomostowe które umożliwiają wyznakowanie aptameru bez utraty jego powinowactwa, jak i zachowanie właściwości fluorescencyjnych barwnika. Odpowiednie znakowanie (np. w miejscu pobliskim obszarowi rozpoznania międzycząsteczkowego) pozwala na śledzenie zmian konformacyjnych aptameru po związaniu docelowej cząsteczki, a sam aptamer pełni rolę biosensora.
Aptamery nukleinowe i przeciwciała
Specyficzność działania aptamerów porównywana jest z przeciwciałami monoklonalnymi. Jednakże oba rodzaje molekuł biologicznie czynnych wykazują zasadnicze różnice (Tab. 1).
Tabela 1. Zestawienie różnic we właściwościach przeciwciał i aptamerów nukleinowych.
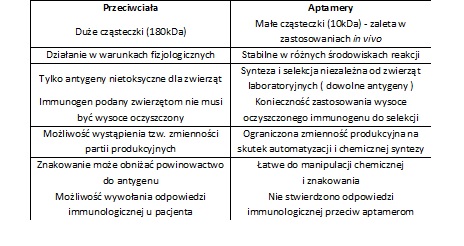
Aptamerami można dość łatwo manipulować chemicznie i znakować bez utraty specyficzności, a ponadto można je otrzymywać w dużej ilości. Specyficzność i selektywność interakcji zachodzącej między aptamerami a wiązanymi przez nie cząsteczkami, pozwala sądzić, że mogą mieć one zastosowanie kliniczne powodując mniej skutków ubocznych niż stosowane obecnie leki.
Zastosowania aptamerów nukleinowych
Techniki immunochemiczne
Test ELONA (ang. enzyme-linked oligonucleotide assay) jest odmianą standardowej techniki ELISA (ang. enzyme-linked immunosorbent assai). Główną różnicą jest zastosowanie obok lub w miejsce przeciwciał specyficznych aptamerów rozpoznających określoną biocząsteczki (najczęściej białko). Aptamery takie mogą być wyznakowane np. fluoresceiną i zastępować przeciwciało wtórne lub też mogą bezpośrednio wiązać docelową cząsteczkę będąc przymocowanym do podłoża i pełniąc rolę przeciwciała pierwotnego. Porównania ELONA z klasycznym testem ELISA wykazały, że jest to także metoda bardzo czuła i precyzyjna.
Radiologia - diagnostyka obrazowa
Radiologia to grupa metod, których celem jest identyfikacja, lokalizacja oraz ocena stopnia zaburzeń w strukturze i funkcji badanych narządów. Metody te wymagają zastosowania bezpiecznych dla organizmu środków kontrastujących o odpowiednim do detekcji sygnale i wysokiej swoistości tkankowej. Taką rolę mogą pełnić wyznakowane aptamery zdolne do wiązania się z białkami występującymi na powierzchni określonych tkanek lub komórek np. zmienionych nowotworowo. Wykorzystując ustalone in vitro linie komórek nowotworowych, udało się wyizolować aptamery skierowane przeciwko tenascynie C (białko markerowe produkowane przez komórki rakowe w dużych ilościach) oraz zewnątrzkomórkowej domenie receptorowej kinazy tyrozynowej RTK (białko transbłonowe zaangażowane w przekazywanie sygnałów koniecznych do proliferacji i wzrostu niektórych nowotworów). Kolejną zaletą zastosowania aptamerów jest możliwość wykorzystania ich do identyfikacji nieznanych markerów komórek nowotworowych określonego typu, co być może umożliwi w przyszłości szybsze wykrycie ognisk rakowych w organizmie, a także monitorowanie przebiegu leczenia in vivo.
Diagnostyka obrazowa z wykorzystaniem nośnika aptamerowego
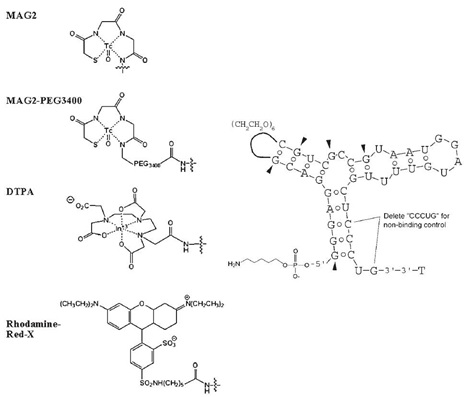
Obecnie aptamery wykorzystuje się w diagnostyce obrazowej jako nośniki barwników fluorescencyjnych takich jak fluoresceina, czy rodamina, które po aktywacji można wykryć technikami spektroskopowymi. Niektóre aptamery stosowane są jako nośniki radionuklidów diagnostycznych, takich jak technet-99m lub ind-111 (Rys. 5).
Terapia celowana z wykorzystaniem nośnika aptamerowego
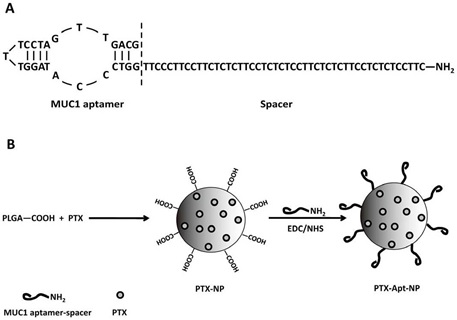
Aptamery stosowane w celowanej terapii antynowotworowej są najczęściej nośnikami nanocząstek polimerowych, które zawierają w swej strukturze leki antymitotyczne typu paklitaksel (ang. paclitaxel, PTX) (Rys. 6). Lek tego typu hamuje podział komórki nowotworowej, przez co jej rozprzestrzenianie zostaje zatrzymane w odpowiedniej fazie.
Aptamer umiejscowiony jest na końcu łącznika (ang. spacer), który oddala go od nanocząstki (eliminacja efektów sterycznych) oraz nadaje całej strukturze odpowiednich właściwości hydrofobowych (rozpuszczalność w błonach cytoplazmatycznych). Takimi łącznikami są najczęściej polimery na bazie glikolu polietylowego (PEG) zakończone wolna grupą aminową. Do tej grupy poprzez wiązanie peptydowe przyłączana jest nanocząstka polimerowa, w której uprzednio zamknięto lek antymitotyczny (PTX-NP).
Najczęściej wykorzystywane aptamery w aplikacjach medycznych
W aplikacjach i badaniach pod kątem zastosowań medycznych skupiono się na aptamerach RNA, które są bardziej „plastyczne” od aptamerów DNA. Istnieje grupa aptamerów, z której wszystkie skierowane są na czynniki zlokalizowane na błonach cytoplazmatycznych komórek nowotworowych. Należy przy tym wspomnieć, że czynniki te występują w dużej nadekspresji tylko i wyłącznie na powierzchni błon komórek nowotworowych. Do powyższej grupy zaliczane są aptamery, takie jak A30, A9, A10, AS-1411, Clone 5, CTLA-4, TTA1, MUC1 oraz PDGF-r.
Podsumowanie
Dynamiczny rozwój zainteresowania aptamerami obserwowany jest od 2002 roku. Ich unikatowe właściwości przejawiające się w specyficzności i selektywności interakcji zachodzącej między aptamerami a wiązanymi przez nie cząsteczkami, pozwalają sądzić, że mogą mieć one zastosowanie kliniczne powodując mniej skutków ubocznych niż stosowane obecnie leki. Jednakże proces dopuszczenia leku do obrotu poprzedzony jest wieloletnimi badaniami klinicznymi. Przez okres dziesięciu lat zaledwie jeden lek na bazie aptameru został dopuszczony do sprzedaży, a w chwili obecnej badaniami klinicznymi objęte jest kolejne osiem [2].
Autor: Karolina Wójciuk
Literatura
2. Esposito, C.L., Catuogno, S., de Franciscis, V., Cerchia, L., 2011. New insight into clinical development of nucleic acid aptamers. Discov Med. 11(61), 487-96.
3. http://www.mpi-inf.mpg.de/departments/d1/projects/CompBio/align.html
4. Tuerk, C., Gold, L., 1990. SELEX: Systematic Evolution of Ligands by Exponential Amplification. Science 249, 505-510.
5. Hicke, B.J., Stephens, A.W., Gould, T., Chang, Y.F., Lynott, C.K., Heil, J., Borkowski, S., Hilger, C.S., Cook, G., Warren, S., Schmidt, P.G., 2006. Tumor targeting by an aptamer. J Nucl Med. 47(4), 668-78.
6. Yu, C., Hu, Y., Duan, J., Yuan, W., Wang, C., Xu, H., Yang, X.D., 2011. Novel aptamer-nanoparticle bioconjugates enhances delivery of anticancer drug to MUC1-positive cancer cells in vitro. PLoS One. 6(9), e24077.









Recenzje