Białka z klastrami żelazowo-siarkowymi
Białka zawierające w swojej strukturze klastry żelazowo siarkowe są szeroko rozpowszechnione. Ich struktura opiera się na skoordynowaniu przez żelazo grup tiolanowych. Budowa ich bywa skomplikowana i zwielokrotniona stąd tez białka te mają różny stopień liczy koordynacyjne, jak i atomów centralnych. Białka te biorą udział w wielu procesach, ze względu na klaster żelazowo siarkowy, są one szczególnie narażone na uszkodzenia oksydacyjne. Reakcje utlenienia powodują inhibicję procesów komórkowych.
Skróty
BER - naprawa struktury DNA z wycięciem zasady (ang, base excision repair)
LIP – labilna pula żelaza (ang. the labile iron pool)
Wprowadzenie
Produkty stresu oksydacyjnego uszkadzają makrocząsteczki (DNA, białka). Jednym z najczęściej pojawiającym się następstwem stresu oksydacyjnego na DNA jest utlenienie jego zasad. Ten typ uszkodzenia powoduje zablokowanie widełek replikacyjnych lub błędnego sparowania zasad, czego wynikiem są mutacje a nawet śmierć komórki. Obecność reaktywnych form tlenu powoduje również zmiany post-translacyjne w strukturze białek (Tab.1)

Tab. 1 Zmiany post-translacyjne wywołane nadmiarem tlenku azotu(II) [ 1]
Postuluje się, ze najbardziej narażone na procesy post-translacyjne są białka zawierające w swojej strukturze żelazo, w tym białka zawierające centra żelazowo-siarkowe [2,3]. Głównym procesem niszczącym strukturę białek z klastrami żelazowo-siarkowego typu [4Fe-4S] jest dysocjacja nitrozylowego kompleksu żelaza [4].
Białka zawierające klaster żelazowo siarkowy
Klastry żelazowo-siarkowe Fe-S są szeroko omawiane w literaturze, ze względu na postulowane znaczenie biologiczne [5]. Szeroko poznane i wykorzystywane są te związki w chemii nieorganicznej jako analogi syntetyczne białek komórkowych. Znanych jest wiele typów białek (Rys. 1).
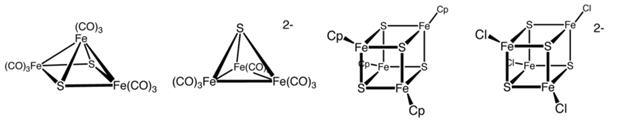
Rys.1 Syntetyczne klastry Fe-S, kolejno: Fe3S2(CO)9, [Fe3S(CO)9]2-, (C5H5)4Fe4S4, [Fe4S4Cl4]2- [6]
W niektórych białkach, pełniących funkcję enzymów, klastry żelazowo siarkowe występują, jako grupy prostetyczne np. enzymy mitochondrialnego łańcucha oddechowego (reduktaza NADH-Q, reduktaza bursztynian-Q, białko Rieske).
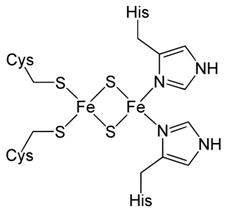
Rys. 2 Centrum żelazowo-siarkowe białka Rieske Centra te mogą również występować w białkach niehemowych, ale zawierających żelazo.
Oznaczenia białek zawierających klaster żelazowo-siarkowy [7]
Znanych jest kilka rodzajów centrów żelazo-siarkowych (Rys. 1). Oznaczenia tych białek są zależne od ilości atomów żelaza i siarki. Najprostszym znanym klastrem jest wiązanie jednego atomu żelaza z czterema resztami cysteiny. Bardziej złożony jest typ drugi określany 2Fe-2S składa się z dwóch jonów żelaza połączonych dwoma atomami siarki nieorganicznej. Jednocześnie każdy z jonów żelaza koordynowany jest zwykle dwoma resztami cysteiny. Trzeci typ centrów 4Fe-4S składa się z czterech jonów żelaza i czterech atomów siarki nieorganicznej połączonych, a struktura przestrzenna tworzy sześcian. Typ 4Fe-4Fe występuje w białku Rieske (Rys. 2).
Wiązanie w klastrach żelazowo- siarkowych [8]
Typowym przedstawicielem tlenowców siarka, posiadającą sześć elektronów walencyjnych. W wyniku przyłączenia dwóch elektronów, tworzą się naładowane, dwuwartościowe jony, które bardzo często reagują z metalami przejściowymi. W wyniku tej reakcji tworzy się wiązanie koordynacyjne pomiędzy atomami siarki a atomami żelaza.. Takie cząsteczki, wchodzą w dalsze reakcje z jonami żelaza tworząc jeszcze bardziej złożone związki kompleksowe.
Anion tiolanowy (RS-) uważany jest powszechnie za jeden z podstawowych ligandów w chemii koordynacyjnej [9]. Jest to ligand o duże gęstości elektronowej stąd też jego powinowactwo do żelaza i innych metali. Siarka tiolowana wykazuje silną tendencję do mostkowania, w rezultacie powstają układy siarczkowe, wielordzeniowe i polimeryczne.
Cysteina jest źródłem swoistego liganda tiolanowego w wielu metaloproteinach, a także w białkach żelazowo-siarkowych [10].
Białko żelazowo-siarkowych - MutY
W celu zapobiegania niekorzystnym wływom reakcji wtórnych stresu oksydacyjnego na makrocząstki w tym DNA jest naprawa z wycięciem zasady (ang, base excision repair, BER). Naprawa ta odbywa się poprzez hydrolizę wiązania N-glikozylowego pomiędzy uszkodzoną zasadą a szkieletem DNA. Do enzymów BER uczestniczących w naprawie należą białka żelazowo-siarkowe np. glikozylazy: MutY, endonukleaza III (EndoIII) oraz uracylowa [11 ,12].
Białko MutY zawiera w swojej strukturze cztery cząsteczki cysteiny z których każda jest źródłem swoistego liganda tiolanowego (Cys-X6-Cys-X2-Cys-X5-Cys).
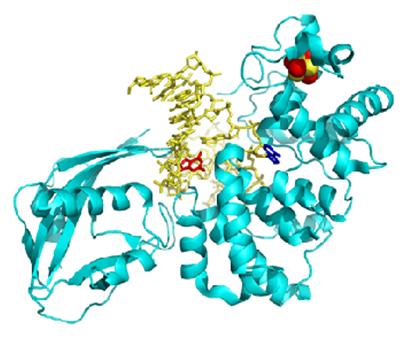
Rys. 2 Struktura białka MutY [13]
Białko MutY ma strukturę drugorzędową. Klaster żelazowo siarkowy nie jest umiejscowiony w części aktywnej enzymu lecz jest oddalony od tego miejsca o około 16 Å.
Białko to może bezpośrednio reagować z reaktywnymi formami tlenu, reakcje te prowadzą do biosyntezy dinitrozylowych kompleksów żelaza. Postuluje się że białko to ma wpływ na LIP (ang. the labile iron pool). Aktywne enzymatycznie białko MutY specyficznie rozpoznaje i przecina nić DNA zawierającą adeninę błędnie sparowaną z 8-oksoguaniną. Przebieg reakcji można sprawdzić wieloma metodami np. metodą elektroforezy na denaturującym żelu poliakrylmidowym. Dzięki zastosowaniu tego białka jest możliwe oznaczenie ilości DNA, które uległo przecięciu w stosunku do całkowitego naniesionego DNA.
Podsumowanie
Białka z klastrami żelazowo-siarkowymi są białkami o skomplikowanej strukturze przestrzennej, mimo to pospolicie występują w komórkach żywych organizmów.
Literatura:
[1] A.R.Butler, F.W.Flitney, D.L.Williams. NO, nitrosonium ions, nitroxide ions, nitrosothiols and iron-nitrosyls in biology: a chemist's perspective, Trends Pharmacol.Sci., 16, (1995) 18-22.
[2] Y.J.Geng, A.S.Petersson, A.Wennmalm, G.K.Hansson. Cytokine-induced expression of nitric oxide synthase results in nitrosylation of heme and nonheme iron proteins in vascular smooth muscle cells, Exp.Cell Res., 214, (1994) 418-428.
[3] J.R.Lancaster, Jr., J.M.Langrehr, H.A.Bergonia, N.Murase, R.L.Simmons, R.A.Hoffman. EPR detection of heme and nonheme iron-containing protein nitrosylation by nitric oxide during rejection of rat heart allograft, J Biol.Chem., 267, (1992) 10994-10998.
[4] E.Soum, J.C.Drapier. Nitric oxide and peroxynitrite promote complete disruption of the [4Fe- 4S] cluster of recombinant human iron regulatory protein 1, J.Biol.Inorg.Chem., 8, (2003) 226-232.
[5] S. J. Lippard, J. M. Berg “Principles of Bioinorganic Chemistry” University Science Books: Mill Valley, CA; 1994
[6] http://www.enotes.com/topic/Iron-sulfur_cluster
[7] Jeremy Mark Berg, John L Tymoczko, Lubert Stryer, Neil D Clarke, Zofia Szweykowska-Kulińska, Artur Jarmołowski, Halina Augustyniak: Biochemia. Warszawa: Wydawnictwo Naukowe PWN, 2007, s. 507-517
[8] The Cambridge Structural Database, CSD ver. 5.29
[9] Comprehensive Coordination Chemistry, tom 2, G. Willkinson (red.) Pergamon Press, Oxford, 1987
[10] S.J. Lippard, J.M. Berg, Podstawy Chemii Bionieorganicznej, PWN, Warszawa 1998
[11] A.K.Boal, E.Yavin, O.A.Lukianova, V.L.O'Shea, S.S.David, J.K.Barton. DNA-bound redox activity of DNA repair glycosylases containing [4Fe-4S] clusters, BiochemistryOK, 44, (2005) 8397-8407.
[12] O.A.Lukianova, S.S.David. A role for iron-sulfur clusters in DNA repair, Curr.Opin.Chem.Biol., 9, (2005) 145-151.
[13] http://www.seongminleelab.com
opracowała: Katarzyna Wójciuk
http://laboratoria.net/home/15995.html








