- Biochemia
- Biofizyka
- Biologia
- Biologia molekularna
- Biotechnologia
- Chemia
- Chemia analityczna
- Chemia nieorganiczna
- Chemia fizyczna
- Chemia organiczna
- Diagnostyka medyczna
- Ekologia
- Farmakologia
- Fizyka
- Inżynieria środowiskowa
- Medycyna
- Mikrobiologia
- Technologia chemiczna
- Zarządzanie projektami
- Badania kliniczne i przedkliniczne
Rybonukleazy: RNaza, RNaza P i onkonaza jako enzymy degradujące kwasy rybonukleinowe (RNA)
Rybonukleazy (RNazy) są enzymami,
których główną funkcją jest rozkładanie wiązań fosfodiestrowych w kwasach
rybonukleinowych (RNA). Obecność tych
specyficznych enzymów stwierdzono u wszystkich organizmów, przy czym w zależności od gatunku różnią się one specyficznością oraz mechanizmem działania
enzymatycznego [1]. Rybonukleazy występują także w skórze ludzkiej, gdzie
niektóre RNazy z ludzkiego naskórka wykazują tylko aktywność hydrolityczną,
podczas gdy inne uważane są za istotne w procesie przyczepności i złuszczania
keratynocytów [6].
Scharakteryzowano dwie główne klasy rybonukleaz, a mianowicie:
- Endorybonukleazy tj. enzymy rozkładające wiązania wewnątrz łańcucha RNA
- Egzonukleazy, których zadaniem jesy uwalnianie nukleotydówz kwasu rybonukleinowego (RNA) na jego zakończeniach.
Jako enzymy rybonukleazy pełnią w komórce różnorodne funkcje. I tak przypisuje się im udział w procesie dojrzewania prekursorów kwasów nukleinowych oraz w rozkładzie RNA. Ponadto, Rnaza I uczestniczy w układzie obronnym organizmu. Choć specyficzność Rnaz wiąże się z jednoniciowymi fragmentami kwasu rybonukleinowego, znana jest też tzw. Rnaza H, która atakuje hybrydy RNA-DNA.
Obok typowych Rnaz występują też tzw. rybonukleazy cyklizujące, które rozkładają wiązania fosfodiestrowe z wytworzeniem pośredniego produktu cyklicznego. Reakcja zachodzi w dwóch etapach:
- Rybonukleazy katalizują specyficzne rozbicie wiązań fosfodiestrowych w kwasach rybonukleinowych. W wyniku ich działania powstają jedno- oraz kilkonukleotydy. W reakcji tej najpierw tworzy się końcowy cykliczny 2’3’-nukleotyd, powstający na skutek przeniesienia reszty fosforowej z węgla 5’ rybozy sąsiedniego nukleotydu na węgiel 2’ rybozy poprzedzającego nukleotydu.
- W drugim etapie dochodzi do hydrolitycznego rozbicia wiązania przy węglu 2’, a następnie tworzy się nukleotyd końcowy, który jest zestryfikowany z kwasem fosforowym przy węglu 3’[1].
W komórkach roślinnych występują rybonukleazy roślinne, które również rozkładają wszystkie rodzaje RNA- najłatwiej jednak atakują mRNA. Aktywność enzymu zależy od bardzo wielu warunków i tak:
- od stanu fizjologicznego komórki
- warunków środowiska (wpływają one na syntezę nowych drobin enzymów, jak również na ich konformację, a to z kolei wpływa na stopień czynności biologicznej)
- aktywność Rnaz zwiększa się w trakcie starzenia się komórki i w ekstremalnych warunkach, co prowadzi do nadmiernego rozkładu RNA oraz zaburzeń w syntezie białek [2].
Ponadto, aktywność RNaz ulega podwyższeniu np. w okresie kiełkowania nasion czy w trakcie zachodzenia procesu apoptozy. Dodatkowo, niektórzy autorzy (np. Lehmann i wsp. 2001, czy Raj i wsp. 2001), donoszą, że do stymulacji aktywności Rnaz dochodzi także podczas ataku fitopatogenów i w warunkach niedoboru fosforu [3].
Udział RNazy P w powstawaniu tRNA
Wszystkie rodzaje tRNA powstają z dłuższych transkryptów tzw. pre-tRNA. Zanim cząsteczki staną się właściwym tRNA przechodzą proces dojrzewania, na który składa się kilka etapów:
- Odcięcie sekwencji liderowej z z końca 5’-
- Odcięcie dwóch nukleotydó z końca 3’
- Dodanie do końca 3’ sekwencji CCA
- Wycięcie centralnego intronu z cząsteczki pre-tRNA.
- Ostatnim etapem dojrzewania jest chemiczna modyfikacja zasad azotowych. Cały ten proces katalizowany jest właśnie przez RNazy oraz inne enzymy, które modyfikują zasady. RNaza P odpowiada za dojrzewanie końca 5’ tRNA.
RNaza P zbudowana jest z jednej cząsteczki RNA (tworzącej centrum ) i jednej cząsteczki białka (odpowiedzialnego za utrzymanie aktywnej konformacji). Jako enzym RNaza P zalicza jest do endonukleaz (podobnie jak RNazy D,E czy F) [4], [5]. Rybonukleaza P(RNaza P) jest więc kluczowym enzymem w biogenezie tRNA, który katalizuje endonukleolityczne rozszczepienie prekursorów tRNA oraz generuje ich dojrzały koniec 5’ [5]. Jedną z pierwszych katalitycznie scharakteryzowanych rybonukleaz była RNaza P pochodząca z bakterii [5].
Izolacja RNazy P z keratynocytów skóry (wg Pavlidou D. i wsp. 2005).
Jako materiał do badań wykorzystano keratynocyty ludzkiej skóry, odpowiednio przygotowane. Komórki pobrano ze skóry pacjentów z otyłością poddanych wcześniej abdominoplastyce (plastyka brzucha) . Komórki przygotowuje się przez odpowiednią preparatykę, na końcu próbki poddaje się wirowaniu w 4C przez 5 minut, a żywotnośc keratynocytó mierzy się testem z błękitem trypanu [6].
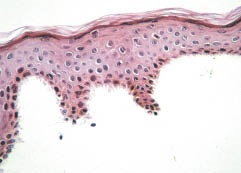
Fig. 1. Complete separation of human epidermis from dermis subsequent to incubation with dispase (haematoxylin-eosin, ×400). Zdjęcie: Kompletny podział ludzkiego naskórka od skóry właściwej po inkubacji z dispazą (naturalna proteaza), http://www.medicaljournals.se/acta/content/?doi=10.2340/00015555-0019&html=1
Oczyszczanie enzymu:
Uzyskane komórki w ilości 5x106 zawieszono w 4 ml lodowatego buforu A (tj. 10 mM HEPES o pH=7.9; 10 mM KCl; 0,1 mM EGTA; 1 mM ditriotreitol (DTT); 0,5 mM fluorek fenylometylosulfonylu (PMSF)) i pozostawiono do spęcznienia na 30 minut. Następnie, dodano (0,6% v/v)detergent Nonidet P-40(octyl phenoxylpolyethoxylethano), po czym zawiesinę homogenizowano, po czym odwirowano. Po wirowaniu otrzymane peletki zawieszono w 1 ml lodowatego buforu C (tj.: 20 mM HEPES o pH=7.9; 0,4 M NaCl; 1mM EDTA; 1mM EGTA; 1mM ditiotreitol; 20% gliceryna), próbkę energicznie wytrząsanow temperaturze 4st.C przez 30 minut. Zanieczyszczenia jądrowe usunięto przez 15 minutowe odwirowanie przy 14000 obr./minutę, po czym ekstrakt przechowywano w -20 st.C [6].
Komórkowe i jądrowe ekstrakty dializowano przez noc w 2 L buforu B (tj.: 50 mM fosforan potasu, pH=7.0; 0,1 mM ditiotreitol; 10% glicerol objetościowo), po czym próbkę w obj. 3 ml naniesiono na kolumnę fosfocelulozową (P-11) zrównoważoną wcześniej buforem B (jak wyżej). Kolumnę pzremywano tym samym buforem do momentu, aż nie zaobserwowano spadku absorbancji A=280 nm do zera. RNazę P eluowano z kolumny za pomocą 9 ml liniowego gradientu od 50 do 500 mM NH4Cl w buforze B. Otrzymane aktywne frakcje enzymu połączono i dializowano przez noc w 2 L buforu K (tj.: 5 mM MgCl2; 100 mM NH4Cl; 50 mM tris-HCl o pH=7.5) w obecności 20% glicerolu. Próbkę podzielono na małe porcje , po czym przechowywano w temperaturze -20 st.C [6].
Aktywność rybonukleolityczna
W homogenatach uzyskanych z komórek zwierzęcych występują dwa optymalne stężenia jonów wodorowych. W pH kwasowym mieszczącym się w granicach 5.2- 5.8 oraz pH zasadowym (7.8 – 8.2) stwierdza się optymalną aktywność RNaz. Co więcej, przeprowadzone badania wykazały, że aktywność tego enzymu w pH kwasowym jest mniejsza, niż w pH zasadowym. Między RNazami kwasowymi i zasadowymi występuje kilka różnić m.in. rybonukleazy kwasowe są wrażliwe na temperaturę i tracą swoją aktywność w rozcieńczonych kwasach, z kolei RNazy zasadowe są stosunkowo odporne na działanie wymienionych czynników [1].
Metoda Mc Donald- izolacja RNazy z tkanki trzustkowej
Metodę otrzymywania krystalicznej rybonukleazy po raz pierwszy opisał Kunitz (1940). Od tego czasu prowadzono badania, w trakcie których wykazano, że enzym ten nie tylko ma zdolność degradowania kwasu rybonukleinowego, ale także do hydrolizy białka [7]. Procedura opisana przez Kunitz’a została nieznacznie zmieniona na przestrzeni lat. W metodyce zmieniono kilka etapów, dzięki czemu udało się otrzymać RNazę wolną od wszelkich zanieczyszczeń, a cząsteczna RNazy jest praktycznie nienaruszona [7].
Metoda izolacji opiera się na ekstrakcji RNazy z tkanki za pomocą 0,12 M roztworu H2SO4. Kolejnym etapem jest frakcjonowanie za pomocą (NH4)2SO4. Całą preparatykę należy wykonywać w temperaturze 4st.C [1].
Otrzymywanie surowego preparatu rybonukleazy
Wykonanie:
Ekstrakcja I: Jako materiał do badań należy wykorzystać trzustkę bydlęcą, z której ekstrahuje się RNazę za pomocą lodowatego 0,12 M roztworu kwasu siarkowego (H2SO4). Cały proces prowadzis ię pzrez 24 godziny, po czym kwasowy ekstrakt pozostawia się w niskiej temperaturze, a trzustkę poddaje się oczyszczaniu (usunięcie tłuszczu oraz tkanki łącznej).
Kolejnym etapem jest homogenizacja tkanki, którą ponownie zalewa się zimnym roztworem H2SO4 (0,12 M) w proporcji: 2 L kwasu na 1 L zmielonej tkanki. Kolejną ekstrakcję prowadzi się przez 18 do 24 godzin, ze sporadycznym wytrząsaniem. Po ekstrakcji, homogenat trzustki należy przesączyć przez gazę, a pozostałą tkankę zalewa się równą objętością 0,12M H2SO4 . Tak przygotowaną próbkę pozostawia się na 1 godzinę [1], [7].
Przesącz otrzymany z trzech kolejnych ekstrakcji należy połączyć, po czym doprowadzić do 65% nasycenia roztworem (NH4)2SO4. W tym celu dodaje się 430 g (NH4)2SO4 w substancji na 1 litr przesączu. W celu uformowania osadu , otrzymany ekstrakt pozostawia się na 24 godziny, po czym odsącza wysolone białka przez sączki wykonane z bibuły Whatman nr 2 [1], [7].
Powstały przesącz zachowuje się, a osad traktuje się równą objętością wody (w stosunku do użytej ilości trzustki bydlęcej) wraz z dodatkiem 430 g (NH4)2SO4 na 1 litr zawiesiny. Tak przygotowaną próbkę ponownie pozostawia się na 24 godziny. Po upływie czasu inkubacji próbkę odsącza się (jak wyżej), a osad odrzuca [1], [7].
Wysolenie surowej frakcji enzymu:
W tym celu połączone przesącze doprowadza się do do 80% nasycenia (NH4)2SO4, dodając 105 gramów (NH4)2SO4 na 1 litr roztworu i odstawia na 24 godziny. Po wytrąceniu osadu ciecz należy ostrożnie zdekantować , a dalej odwirować przy 400 obr./minutę w wirówce z chłodzeniem. Otrzymany w ten sposób osad stanowi surowy preparat RNazy [1], [7].
Oczyszczanie RNazy
W celu oczyszczenia preparatu RNazy z enzymów proteolitycznych, należy wcześniej otrzymany osad enzymu rozpuścić w wodzie w proporcji 5 ml wody na 1 g osadu. Ponadto, do roztworu enzymu należy dodać 20% nasycony roztwór (NH4)2SO4 (pH=3) z wyliczeniem 20 ml roztworu (NH4)2SO4 na 1 g RNazy. Roztwór (NH4)2SO4 doprowadza się do odpowiedniego pH za pomocą 5M roztworu kwasu siarkowego.
Tak przygotowaną próbke podgrzewa się do temperatury w granicach 95 st.C - 100 st.C stale mieszając, po czym próbkę gwałtownie się oziębia do temperatury ok. 20-25 st.C. Następnie, poddaje się próbkę 1-godzinnej inkubacji (w tej temperaturze). W trakcie inkubacji dochodzi do wytrącenia się enzymó proteolitycznych, które następnie adsorbowane są na ziemi okrzemkowej (1 g ziemi okrzemkowej na 100 ml zawiesiny). Osad pzresącza sie na lejku Buchnera, a następnie przemywa się 3-krotnie za pomocą małych ilości zakwaszonego do pH=3 roztworu (NH4)2SO4 (20% nasycenie). W dalszym etapie oczyszczania, otrzymany przesącz doprowadza się do dp 50% nasycenia (NH4)2SO4 (dodając 18,8 g (NH4)2SO4 na 100 ml cieczy). Po upływie godzinnej inkubacji, osad należy odsączyć, po czym go odrzucić. RNazę odzyskuje się z przesączu po wysoleniu za pomocą siarczanu amonu o 80% nasyceniu ( w tym celu dodaje się 21 g (NH4)2SO4 na 100 ml przesączu). Po upływie 24 godzin należy odwirować osad enzymu [1], [7].
Krystalizacja jest metodą oczyszczania związków chemicznych. Cały proces polega na otrzymaniu nasyconego roztworu substancji, którą oczyszczamy w temperaturze wrzenia rozpuszczalnika. Następnie, roztwór poddaje się sączeniu (oddzielenie ewentualnych nierozpuszczalnych zanieczyszczeń) i ochłodzeniu (otrzymanego przesączu) [11].
Zazwyczaj, stałe związki organiczne, które są bezpośrednio wydzielone w określonej reakcji nie są chemicznie czyste (zawierają niewielkie ilości innych związków określanych mianem zanieczyszczeń). Zanieczyszczenia powstają jednocześnie z głównym produktem reakcji, w związku z czym próbki takie wymagają oczyszczenia. W większości przypadków oczyszcza się je przez krystalizację z odpowiednim rozpuszczalnikiem lub z mieszaniną rozpuszczalników.
Sam proces krystalizacji polega na wykorzystaniu różnicy rozpuszczalności związków w odpowiednich rozpuszczalnikach (lub ich mieszaninach) [12].
Do podstawowych etapów krystalizacji zalicza się:
- rozpuszczenie zanieczyszczonej substancji w odpowiednio dobranym rozpuszczalniku- proces ten zachodzi w temperaturze wrzenia rozpuszczalnika (lub w jej pobliżu)
- odsączenie gorącego roztworu (od nierozpuszczalnych składników oraz zanieczyszczeń)
- ochłodzenie przesączu (krystalizacja rozpuszczonego związku)
- oddzielenie kryształów od roztworu (tzw. ługu pokrystalicznego)
- wysuszenie otrzymanego związku stałego
- określenie czystości związkku [12].
Przygotowanie RNazy do krystalizacji
Otrzymany czysty preparat RNazy (oczyszczony z enzymów proteolitycznych) w ilości 1 g rozpuszcza się w 5 ml wody, po czym za pomocą 5M roztworu wodorotlenku sodu doprowadza się go do pH=4.8. Po uzyskaniu pożądanego pH do próbki dodaje się 5 ml nasyconego roztworu siarczanu amonu (na 1 g enzymu dodaje się 1 g ziemi okrzemkowej na 100 ml cieczy). Osad odsącza się i odrzuca, zaś przesącz doprowadza się do niższego pH=4.2 (0,5 M roztworem kwasu siarkowego – H2SO4) i wysala RNazę, doprowadzając do 70% nasycenia siarczanem amonu przez dodanie 67 ml nasyconego roztworu (NH4)2SO4 na 100 ml przesączu. Po upływie 24 godzin inkubacji w 4 st.C osad rybonukleazy należy odwirować przy 400 obr./minutę [1], [7].
Krystalizacja RNazy z siarczanem amonu
Całą preparatykę należy prowadzić w temperaturze 0- 4 st.C .
Enzym przygotowany do krystalizacji rozpuszcza się w 1 ml wody ( w stosunku 1g enzymu : 1 ml H2O). Występujące w próbce substancje zanieczyszczające adsorbuje się na ziemi okrzemkowej dodanej w stosunku: 5 g ziemi okrzemkowej na 100 ml roztworu. Powstały osad odsącza się na bibule, po czym przemywa się za pomocą wody.
Następnie, otrzymany przesącz należy doprowadzić do pH równego 4.2- w tym celu stosuje się 1 M roztwór wodorotlenku sodu (NaOH) lub 0,5 M roztwór kwasu siarkowego (H2SO4)- próbkę należy delikatnie mieszać, aż do momentu pojawienia się opalizującego zmętnienia roztworu. Proces krystalizacji należy przeprowadzać w cieplarce o temp. 20-25 st.C. Po upływie 3 dni, powstały osad odwirowuje się, a ług pokrystalizacyjny doprowadza się ponownie do pH równego 4.2 i znów krystalizuje (jak wyżej) [1], [7].
Proces rekrystalizacji
Krystaliczną RNazę rozpuszcza się wodzie, stosując proporcję: 2 ml wody na 1 g otrzymanego skrystalizowanego enzymu. Do rozpuszczonej próbki dodaje się 0,2 g ziemi okrzemkowej (na 2 ml roztworu), a całośc odsącza się na bibule. Pozostały po sączeniu osad (znajdujący się na sączku) należy przemyć małymi porcjami wody, a następnie doprowadzić do pH=4.2 (dodaje się nasyconego roztworu (NH4)2SO4 do momentu pojawienia się delikatnej opalescencji w próbce. Taką próbkę krystalizuje się ponownie (w temp. 20-25 st.C) prze 48 godzin. Otrzymany krystaliczny osad poddaje się wirowaniu, zaś otrzymany po wirowaniu supernatant doprowadza się ponownie do pH=4.2. Supernatant dalej krystalizuje się przez okres 3 dni (jak wyżej) [1], [7].
Rekrystalizowaną Rnaze można również rozpuścić w wodzie (w 1,5 ml) i dializować wobec wody. Cały proces należy prowadzić w temperaturze 0-4 st.C przez 24 godziny. Do dializatu w temp. 5 st.C , dodaje się następnie 96% roztwór etanolu (w stosunku: 12 ml etanolu na 1 ml roztworu). Dochodzi do formowania się osadu (w trakcie odstania próbki przez 2 dni w temperaturze 10 do 20 st.C). Odstany po czasie inkubacji krystaliczny osad, należy przemyć kilka razy 96% etanolem, po czym wysuszyć w eksykatorze nad CaCl2. Opisany proces nazywa się krystalizacją z etanolu [1], [7].
Onkonaza - białko z aktywnością rybonukleazy
Onkonaza (ONC) jest stsounkowo niedawno poznanym białkiem, które otrzymuje się z jaj żaby Rana pipiens. Jest białkiem o masie cząsteczkowej rónej 12000D, a buduje je pojedynczy łańcuch polipeptydowy składający się ze 104 aminokwasów. Onkonaza podobnie jak inne RNazy wykazuje wysoki stopień powinowactwa do niektórych RNaz występujących w organizmie człowieka (np. do RNazy trzustkowej czy wątrobowej)[8].
Przeprowadzone badania wykazały, że białko spełnia szereg bardzo ważnych funkcji. Działanie biologiczne tego białka, a zwłaszcza przeciwnowotworowe, jest ściśle związane z aktywnością rybonukleazy. Onkonaza prowadzi do śmierci komórki, przez rozkład wewnątrzkomórkowego RNA, ponadto odpowiada za hamowanie syntezy białek, a także wzrostu i proliferacji. Ponadto, wykazano, że odpowiada za selektywną indukcję apoptozy komórek nowotworowych (programowana śmierć). Ważnym elementem jej działania cytotoksycznego jest również aktywność antyoksydacyjna [8]. Podobnie jak inne RNazy, onkonaza ma zdolność rozkładania ds RNA (jednoniciowego RNA). Może również tworzyć aktywne biologicznie dimery.
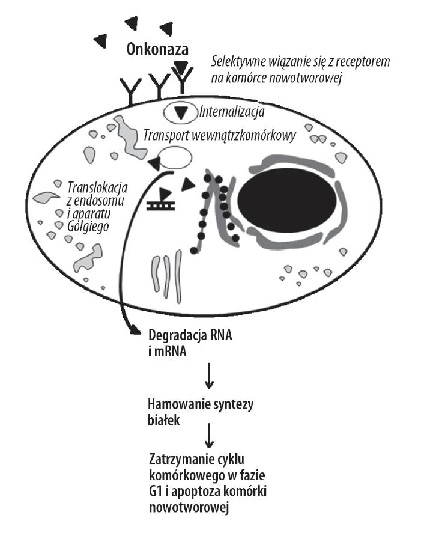
Zdjęcie: Mechanizm działania białka onkonazy [8]. http://www.phmd.pl/fulltxthtml.php?ICID=905228
Onkonaza uważana jest za najmniejsze białko rodziny RNaz A, które zostało wyizolowane z oocytów lub wczesnych embrionów żaby (Rana pipiens). Onkonaza jest białkiem stabilnym (temperatura denaturacji) nawet w temperaturze sięgającej 87 st.C, ponadto jest odporna na degradację przez różne proteazy. Białko to indukuje apoptozę komórek nowotworowych, a samo posiada niską cytotoksyczność [9].
W przeprowadzonych badaniach Saxena S. K. i wsp. (2003), stwierdzili, że onkonaza jest mniej wydajnym enzymem niż RNaza A. W optymalnych warunkach onkonaza wykazała się kilkaset razy niższą szybkością reakcji w porównaniu do RNazy A. Optymalnym pH działania dla onkonazy okazało sie pH=6.0, w którym to onkonaza wykazuje mniejszą różnicę szybkości reakcji w porównaniu do RNazy A [10].
Autor: Lidia Koperwas
Literatura:
[1]. Kłyszejko-Stefanowicz L, 2003. Ćwiczenia z biochemii. Wydawnictwo Naukowe PWN, 2003, s.544-548
[2]. Dolnicki A., 1979. Wpływa stymulatoró wzrostu na aktywność rybonukleaz i na metabolizm kwasów rybonukleinowych u roślin. Wiadomości botaniczne Tom XXIII - zeszyt 3, 1979.
[3]. Sytykiewicz H., Czerniewicz P., Leszczyński B., Sempruch C., Goławska S., Sprawka I., 2011. Aktywność rybonukleolityczna w liściach Czeremchy Zwyczajnej w okresie żerowania mszycy czeremchowo-zbożowej (Rhopalosipium Padi L.). Progress in Plant protection /Postępy w Ochronie Roślin 51 (1) 2011. http://www.progress.plantprotection.pl/pliki/2011/PPP_51_1_29_Sytykiewicz_H.pdf
[4]. Żak I. Kwasy nukleinowe, rozdział 19. http://biochigen.sum.edu.pl/podrecznik/19.pdf
[5]. Kudła M. Rybozymy. RNA- nośnik informacji i narzędzie katalizy enzymatycznej. http://www.igib.uw.edu.pl/~knbm/pdf/rybozymy.pdf
[6]. Pavlidou D., Vourekas A., Monastirli A., Kalavrizioti D., Tsambaos D., Drainas D., 2005. Isolation of Ribonuclease P Activity From Human Epidermis and its Regulation by Retinoids In vitro. Acta Derm Venereol 2006; 86: 114–118. http://www.medicaljournals.se/acta/content/?doi=10.2340/00015555-0019&html=1
[7]. McDonald M.R., 1948. A method for the preparation of “protease-free” crystalline ribonuclease. The Journal of General Physiology. http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2213746/pdf/39.pdf
[8]. Zwolińska M., Smolewski P., 2010. Onkonaza-rybonukleaza o aktywności przeciwnowotworowej. Postepy Hig Med Dosw. (online), 2010; 64: 58-66. http://www.phmd.pl/fulltxthtml.php?ICID=905228 [9]. Meng Qiao, Li-Dong Zu, Xiao-Hong He, Ru-Ling Shen, Qing-Cheng Wang, ,Mo-Fang Liu , 2012. Onconase downregulates microRNA expression through targeting microRNA precursors. Cell Research (2012) 22:1199–1202. doi:10.1038/cr.2012.67; published online 24 April 2012. http://www.nature.com/cr/journal/v22/n7/full/cr201267a.html
[10]. Saxena S.K., Shogen K., Ardelt W., 2003.Alfacell Corporation, Bloomfield, NJ. Onconase and it’s therapeutic potential. Laboratorymedicine, may 2003. Number 5, volume 34. http://labmed.ascpjournals.org/content/34/5/380.full.pdf
[11]. http://www.e-biotechnologia.pl/Artykuly/krystalizacja
[12]. http://biomist.pl/chemia/artykuly/krystalizacja/43
Tagi: RNaza, rybonukleazy, RNaza P, tRNA, keratynocyty skóry, aktywność rybonukleolityczna, metoda McDonald, oczyszczanie, preparat, enzym, onkonaza, wysolenie, rozkład, kwas rybonukleinowy, rekrystalizacja, krystalizacja, etanol
wstecz Podziel się ze znajomymi









Recenzje