- Biochemia
- Biofizyka
- Biologia
- Biologia molekularna
- Biotechnologia
- Chemia
- Chemia analityczna
- Chemia nieorganiczna
- Chemia fizyczna
- Chemia organiczna
- Diagnostyka medyczna
- Ekologia
- Farmakologia
- Fizyka
- Inżynieria środowiskowa
- Medycyna
- Mikrobiologia
- Technologia chemiczna
- Zarządzanie projektami
- Badania kliniczne i przedkliniczne
Bakteriocyny jako alternatywa dla chemicznych konserwantów
Izolacja genomowego DNA
Przed wykonaniem izolacji, hodowlę przeszczepiono i inkubowano w 37°C, przez dobę. Po całonocnej hodowli, pobrano 3 ml i zwirowano z prędkością (8000 rpm/4°C/5 min). Następne etapy były przeprowadzane na lodzie. Izolację DNA wykonano za pomocą mieszanin, których głównym składnikiem był fenol i chloroform, według metody odczynnikowej (SENGÜVEN I IN., 2014). Izolację wykonano dwukrotnie.
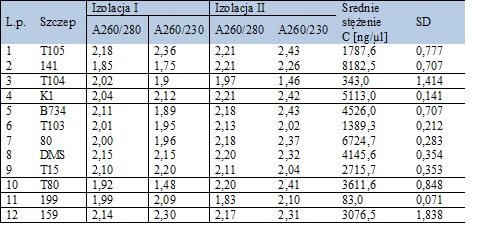
Bezpośrednio po izolacji DNA wykonano analizę jakościową i ilościową materiału, z użyciem spektrofotometru NanoDrop, mierzącym absorbancję w przedziale od 230 do 320 nm. Wyzerowano spektrofotometr wodą dejonizowaną, w której zostało zawieszone DNA. Następnie naniesiono pipetą 1μL roztworu DNA na spektrofotometr, pobranego kolejno z probówek trzymanych na lodzie. Po każdym pomiarze wyczyszczono okienko aparatu. Stosunek absorbancji A260/A280, wynoszący od 1,7-2,0, wskazuje na wystarczające oczyszczenie preparatu. Do dalszych analiz wybrano próbki o najlepszej czystości oraz sporządzono rozcieńczenia 50 ng/µl.
PCR
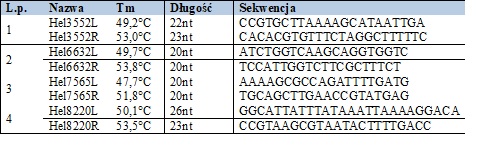
Z wykorzystaniem sekwencji helwetycyny J, zdeponowanej w NCBI GenBank, zaprojektowano startery specyficzne do genu kodującego helwetycyne J (Tab. 1). Startery zaprojektowano tak, aby amplikony nie nachodziły na siebie oraz aby można było złożyć cały operon helwetycyny J, o długości 3300 nt. Nazwa starterów to analogicznie: hel1- liczba porządkowa, 552- to długość produktu danej pary starterów, L, R – położenie na amplikonie w konfiguracji 5’-3’.
Tabela 1. Startery.
Pelet oligonukleotydów, zwirowano, zalano odpowiednią ilością 10 mM Tris-HCl o pH 8, pozostawiono na 3 minuty, a następnie zworteksowano. Tak przygotowane roztwory wyjściowe starterów, rozcieńczano do reakcji w proporcjach: 2 µl startera + 18 µl buforu. Do reakcji użyto DreamTaq PCR Master Mix (2X) (Thermo Scientific), zawierające polimerazę DreamTaq DNA, zoptymalizowany DreamTaq bufor, MgCl2 oraz dNTP. Składniki reakcji PCR na jedną próbkę:12 µl Master Mix, po 1 µl startera R i L, 1 µl materiału DNA, 7 µl wody wolnej od nukleaz. Do każdej pary starterów warunki reakcji zostały zoptymalizowane, poprzez zmiany w liczbie cykli reakcji, zmiany w temperaturze przyłączania starterów, czy wydłużeniu czasu elongacji. Dla Hel3552L, Hel3552R wykonano 30 cykli składających się na: denaturacja początkowa 94°C przez 5 minut, denaturacja właściwa 94°C przez 30 sekund, przyłączanie starterów 57°C przez 30 sekund, elongacja 72°C przez 2 minuty oraz wydłużanie końcowe 72°C przez 10 minut. Analogicznie dla Hel6632L, Hel6632R 94°C – 5 min, 94°C – 30 s, 48,7°C – 30 s, 65°C – 3 min, 65°C – 3 min, dla Hel7565L, Hel7565R 94°C – 10 min, 94°C – 30 s, 46°C – 30 s, 72°C – 2 min, 72°C – 7 min, dla Hel8220L, Hel8220R 94°C – 10 min, 94°C – 30 s, 49°C – 30 s, 72°C – 2 min, 72°C – 10 min. Reakcje przebiegały w termocyklerze (SensoQuest labcycler).
Rozdział elektroforetyczny
Po przeprowadzeniu reakcji PCR, próbki rozdzielano na żelu agarozowym 1%, z dodatkiem bromku etydyny. Elektroforezę prowadzono przy napięciu 80V, przez 1h 30 minut. Produkty reakcji PCR nakładano na żel wraz z buforem obciążającym DNA Gel Loading Dye (6x) (Thermo Scientific) oraz z markerem GeneRuler 1 kb Plus DNA Ladder (Thermo Scientific).
Sekwencjonowanie
Po wizualizacji prążków pod światłem UV, otrzymane sekwencje nukleotydowe oznaczano stosując BigDye Terminator v3.1 Cycle Sequencing Kit (Applied Biosystems, USA) oraz kapilarny system sekwencjonowania 3730 Genetic Analyzer (Applied Biosystems, USA). Sekwencje DNA opracowano za pomocą programu ClastalW oraz programu MEGA 5.0.
Wyniki badań
W wyniku izolacji metodą odczynnikową, wykonano pomiar czystości i jakości materiału genomowego w dwóch powtórzeniach. W tabeli nr 2 przedstawiono średnie wyniki z obu izolacji, uzyskane za pomocą spektrofotometru NanoDrop, mierzącym absorbancję w przedziale od 230 do 320 nm.
Tabela 2. Wyniki izolacji.









Recenzje